サービス概要
DNAメチル化は、DNA配列の変化を伴わずに、様々な生命現象の遺伝子発現や表現型に関わる制御機構です。
DNAメチル化は、CpGアイランドと呼ばれるシトシンの次にグアニンが現れるタイプの2塩基配列の部分などにメチル基が付加される化学反応であり、エピジェネティクスに深く関与しています。
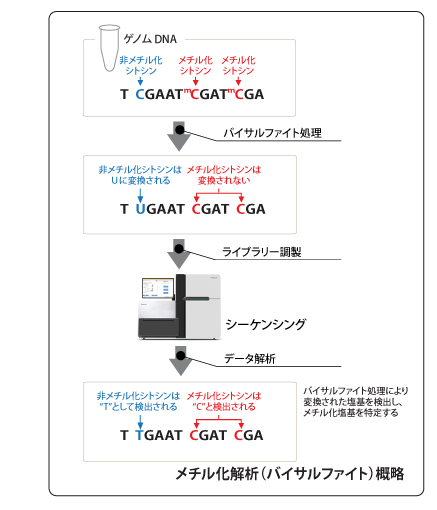
このDNAのメチル化解析を、バイサルファイトシーケンスにより実施しています。
ゲノムDNAに対してバイサルファイト処理を行うと、メチル化シトシンは変換されず、非メチル化シトシンがウラシルに変換されます(シーケンス反応ではウラシルはチミンとして検出されます)。このようにメチル化シトシンが変換されないことを利用し、処理前後のシトシン/ウラシル(チミン)の配列差を見分けることで、塩基レベルでメチル化の有無や部位を精度良くゲノムワイドに解析します。
また、各塩基におけるメチル化の有無の比率をリード数から算出することで、メチル化修飾率を定量的に解析することも可能です。
アプリケーション例
イルミナ社のショートリードのシーケンサー(NovaSeq X Plus)を用いて、低コストに大量のデータを取得します。
取得データ量の目安は、ゲノムサイズの 50-60 倍程度のデータを取得することが推奨されております。
ヒト・マウスの場合、ゲノムサイズが 約3 Gb になりますので、150 Gb/sample のデータを取得がお勧めになります。
代表的な仕様
- ライブラリ調製:Accel-NGS® Methyl-Seq DNA Library Kit
- シーケンス条件:NovaSeq X Plus, 150 bp, paired end, 数十~数百 Gb /sample
- データ解析:Mapping, Peak call, 群間比較(Differential methylation region)
納期
シーケンスのみご依頼の場合:サンプル受領から約 4-6 週間
データ解析までご依頼の場合:サンプル受領から約 8 週間
サンプル受領のタイミングや取得したデータによっては、納期が前後する場合がありますので、ご了承ください。
ご依頼の流れ

オーダーシート(依頼書)をダウンロード
発送内容確認書をダウンロード
FAQ
品質チェックや納品物に関するよくあるご質問については、こちらをご覧ください。
お問い合わせ先
フォームからのお問い合わせは、下記のお問い合わせボタンより、必要事項をご入力の上、送信ください。
メールでのお問い合わせは、bio@apro-s.com までご連絡ください。
お電話でのお問い合わせは、088-678-6372 までご連絡ください
